
myBaits Custom
设计简便–可以以多种来源核酸设计探针 - 基因组,转录组,SNPs
科学家团队–来自专家团队的免费探针设计与项目技术支持
试剂盒有多种规格–Panel和试剂盒规格可以适应多种规模的项目
- 产品简介
- 产品应用
- 引用文献
myBaits 定制试剂盒可为多种物种和规模的实验项目提供NGS杂交捕获panel。客户根据特异性和对目标序列的杂交灵敏度来选择探针。myBaits试剂盒已成功地应用到广泛的目标生物中,如动物、植物和微生物,样本可以来源于新鲜DNA、降解DNA和环境DNA。
本试剂盒能够帮助实验者容易的评估出适用于NGS的多种类型的遗传特征,如点突变,拷贝数变异(CNV),大/小片段插入缺失等等。此外,目标序列捕获适用于多种类型标本的捕获,甚至是非常短的、已降解的样本,如古DNA、法医DNA或cell free DNA。myBaits定制目标捕获试剂盒兼容主要的NGS平台(Illumina®,Ion Torrent®,PacBio®和Nanopore®),并且同一个探针套装在短测序和长测序中皆适用。
产品特点:
设计简便–可以以多种来源核酸设计探针 - 基因组,转录组,SNPs
科学家团队–来自专家团队的免费探针设计与项目技术支持
试剂盒有多种规格–Panel和试剂盒规格可以适应多种规模的项目
完整的实验用组分–试剂盒中包含探针以及用于杂交和洗涤的试剂
产品列表(其他规格请联系我们获取)
| 货号 | 产品名称 | 规格 | 适用样本数量* |
| 300116.v5 | Designs With 1-20K Probes (~1 Mb) | 16 Rxn | 128 |
| 300196.v5 | Designs With 1-20K Probes (~1 Mb) | 96 Rxn | 768 |
| 300516.v5 | Designs With 80-100K Probes (~5 Mb) | 16 Rxn | 128 |
| 300596.v5 | Designs With 80-100K Probes (~5 Mb) | 96 Rxn | 768 |
| 300116M.v5 | Designs With 1-20K Probes | 16 Rxn | 128 |
| 300196M.v5 | Designs With 1-20K Probes | 96 Rxn | 768 |
| 300516M.v5 | Designs With 80-100K Probes | 16 Rxn | 128 |
| 300596M.v5 | Designs With 80-100K Probes | 96 Rxn | 768 |
*依据高质量基因组DNA样本和短读长测序估算的样本量。其他情况请参考 myBaits v5 manual。
myBaits Custom DNA-Seq
Customized NGS Target Capture Kits for DNA Samples
myBaits Custom DNA-Seq杂交捕获探针和试剂可以从NGS DNA文库中快速、特异性地富集目标区域。myBaits Custom DNA-seq探针使用Arbor独特的oligo合成技术合成,与Arbor优化的“v5”试剂搭配,在各种应用中都能有出色的富集性能,且物种和规模不限。
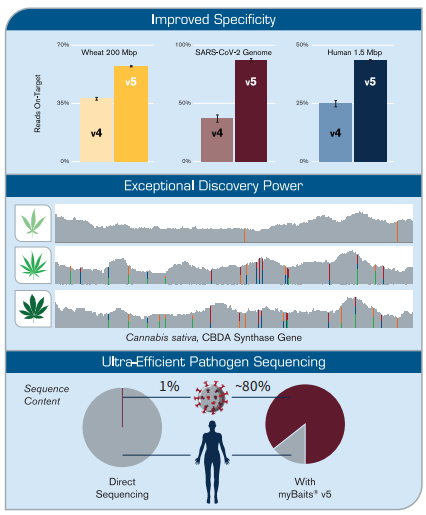
新版myBaits“v5”杂交捕获技术实现了更高的on-target率和灵敏度,可用于发现特殊突变体。例如,从高度复杂的宿主或环境DNA样本中高效地测序完整病原体基因组。
myBaits Custom RNA-Seq
Customized NGS Target Capture Kits for RNA Samples
myBaits Custom RNA-seq杂交捕获探针和试剂可以从NGS RNA文库中快速、特异性地富集目标区域。全转录组测序是一种直接评估基因表达的强大技术,但完全解析信号通常需要对样本进行高深度测序,尤其是罕见的转录本。myBaits Custom RNA-Seq杂交捕获试剂盒,可以为您感兴趣的基因定制探针,富集RNA-Seq文库中的目标序列,并保留转录丰度的相对信号,大大降低NGS的成本。
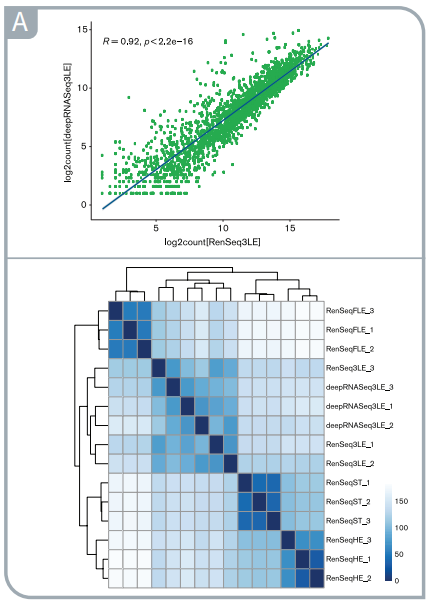
使用 myBaits Custom RNA-Seq 捕获试剂盒富集 RNA-Seq 文库的结果准确且重复性高。
(A)比较每个位点的覆盖范围,此图表明了捕获试剂盒的富集效果和总 RNA-Seq文库之间高度相关。
(B)不同组织中基因表达的层次聚类图。
myBaits Custom Methyl-Seq
Customized NGS Target Capture Kits for Methylation Sequencing
myBaits Custom Methyl-Seq 杂交捕获探针和试剂可以从亚硫酸氢盐或酶转化处理的 NGS DNA 文库中快速、特异性地富集目标区域。甲基化测序是一种直接评估基因组表观遗传修饰的强大技术,但对于感兴趣的位点,直接对样本进行高深度的甲基化测序,费用高,效率低。 myBaits Custom Methyl-Seq 杂交捕获试剂盒,使用独特的探针设计算法,针对您感兴趣的基因提供高灵敏度的定制探针,富集 Methyl-seq 文库中的目标序列,且保留序列中的甲基化信号。与 WGBS 相比, myBaits Custom Methyl-Seq 杂交捕获试剂盒在提高目标覆盖率的同时,大大降低了测序成本,且比扩增子测序更通用、更高效。

Arbor 采用创新探针设计方法,模拟两条转化基因链上不同甲基化状态进行探针设计,并针对目标特异性进行探针过滤,以最大限度地减少脱靶。myBaits Custom Methyl-Seq系统通过对富集参数和化学方法的优化来提高捕获效率。
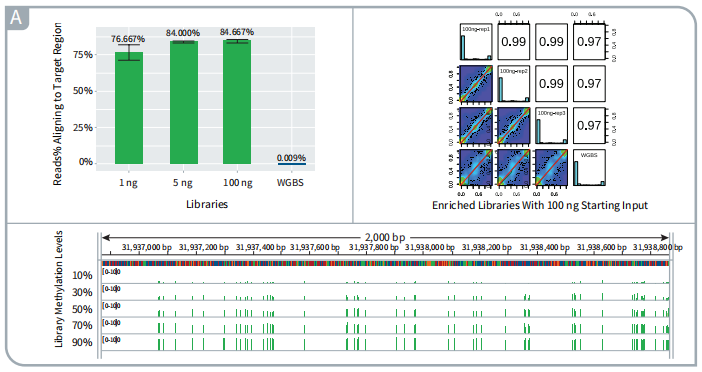
使用 myBaits Custom Methyl-Seq 杂交捕获试剂盒富集methyl-seq文库的结果准确、灵敏、高效、可重复。
(A)与 WGBS 相比,目标区域覆盖率高,节省了大量的测序成本。
(B)富集方法测定的CpG甲基化水平在不同重复之间显示出高度的一致性,并重现了WGBS测定的CpG甲基化水平结果。
(C)IGV截图显示,在不同甲基化水平的捕获文库中,STK12启动子基因座的甲基化水平均匀分布。
Colson, P., C. Dhiver, C. Tamalet, J. Delerce, O.O. Glazunova, M. Gaudin, A. Levasseur, D. Raoult. (2020). Dramatic HIV DNA degradation associated with spontaneous HIV suppression and disease-free outcome in a young seropositive woman following her infection. Scientific Reports 10, 2548
Forth, J.H., L.F. Forth, J. King, O. Groza, A. Hübner, A.S. Olesen, D. Höper, L.K. Dixon, C.L. Netherton, T.B. Rasmussen, S. Blome, A. Pohlmann, M. Beer. (2019). A Deep-Sequencing Workflow for the Fast and Efficient Generation of High-Quality African Swine Fever Virus Whole-Genome Sequences. Viruses. 2019; 11(9):846
Guitor, A.K., A.R. Raphenya, J. Klunk, M. Kuch, B. Alcock, M.G. Surette, A.G. McArthur, H.N. Poinar, G.D. Wright. (2019). Capturing the Resistome: a Targeted Capture Method To Reveal Antibiotic Resistance Determinants in Metagenomes. Antimicrobial Agents and Chemotherapy 64(1): e01324-19
Furtwängler, A., J. Neukamm, L. Böhme, E. Reiter, M. Vollstedt, N. Arora, P. Singh, S.T. Cole, S. Knauf, S. Calvignac-Spencer, B. Krause-Kyora, J. Krause, V.J. Schuenemann, A. Herbig. (2020). Comparison of target enrichment strategies for ancient pathogen DNA. BioTechniques 2020 69:6, 455-459
Knauf, S., J.F. Gogarten, V.J. Schuenemann, H.M. De Nys, A. Düx, M. Strouhal, L. Mikalová, K.I. Bos, R. Armstrong, E.K. Batamuzi, I.S. Chuma, B. Davoust, G. Diatta, R.D. Fyumagwa, R.R. Kazwala, J.D. Keyyu, I.A.V. Lejora, A. Levasseur, H. Liu, M.A. Mayhew, O. Mediannikov, D. Raoult, R.M. Wittig, C. Roos, F.H. Leendertz, D. Šmajs, K. Nieselt, J. Krause, S. Calvignac-Spencer. (2018.) Nonhuman primates across sub-Saharan Africa are infected with the yaws bacterium Treponema pallidum subsp. pertenue. Emerging Microbes & Infections 7:1, 1-4
Ross, Z.P., J. Klunk, G. Fornaciari, V. Giuffra, S. Duchêne, A.T. Duggan, D. Poinar, M.W. Douglas, J-S. Eden, E.C. Holmes, H.N. Poinar. (2018). The paradox of HBV evolution as revealed from a 16th century mummy. PLOS Pathogens 14(1): e1006750
Shipley, M.M., M.M. Rathbun, M.L. Szpara. (2020). Oligonucleotide Enrichment of HSV-1 Genomic DNA from Clinical Specimens for Use in High-Throughput Sequencing. Herpes Simplex Virus – Methods in Molecular Biology Book Series.








